https://www.scieneers.de/wp-content/uploads/2025/02/Output-desinfonavigator_0.png
914
1672
Nico Kreiling
https://www.scieneers.de/wp-content/uploads/2020/04/scieneers-gradient.png
Nico Kreiling2025-02-21 11:03:012025-02-26 11:07:03DesinfoNavigator
https://www.scieneers.de/wp-content/uploads/2025/02/Output-desinfonavigator_0.png
914
1672
Nico Kreiling
https://www.scieneers.de/wp-content/uploads/2020/04/scieneers-gradient.png
Nico Kreiling2025-02-21 11:03:012025-02-26 11:07:03DesinfoNavigatorErforschung des Dark Genome mit Machine Learning zur Entwicklung neuartiger Krankheitsinterventionen
Forschung & Entwicklung
Seltene Erkrankungen sind selten, aber selten sind viele.
350 Millionen, fast 5% der Weltbevölkerung leben mit einer seltenen Erkrankung. Etwa 75% der seltenen Erkrankungen betreffen Kinder. 80% dieser Erkrankungen entstehen durch eine einzige genetische Veränderung und können durch eine Genomanalyse diagnostiziert werden. Das menschliche Genom besteht aus etwa 3.3 Milliarden Bausteinen und jeder Mensch trägt etwa 3.5 Millionen Varianten. Die Suche nach der einen, pathogenen Variante, als Ursache der Krankheit, gleicht der Suche nach der Nadel im Heuhaufen.
Wie kann Maschinelles Lernen und Cloud Computing bei der Suche helfen?
Alle PatientInnen mit einer seltenen Erkrankung, und dies sind in ihrer Gesamtheit vier Millionen Menschen in Deutschland, sowie Personen mit einer Krebserkrankung – jährlich 500.000 Neuerkrankungen – werden in nächster Zukunft in der Routinediagnostik eine umfassende genetische Untersuchung erhalten. Treiber dieser Entwicklung sind die sinkenden Kosten für Genomanalysen, die mit neuen Technologien wie dem Next-Generation Sequencing (NGS) schnell und effizient durchgeführt werden können.
Damit steht die moderne Medizin an der Schwelle zu einer datengetriebenen Disziplin, die die Krankenversorgung nachhaltig verändert. Mehr und mehr rücken molekulare Daten von PatientInnen in den Fokus und ermöglichen die Diagnosestellung, erlauben Prognosen und verbessern Therapie und Prävention.
Der Mensch ist bislang nicht in der Lage, das unmittelbar therapeutische Potential der computergenerierten Daten vollumfänglich zu nutzen und in die Klinik zu übersetzen. Mit bereits unterstützenden bioinformatischen Lösungen zur Genomauswertung lässt sich das Potential bislang immer noch nur im unteren einstelligen Prozentbereich ausschöpfen. Dies liegt vor allem an der unzureichenden Auswertbarkeit von über 90% des Genoms (“Dark Genome”). Neue Analysestrategien werden dringend benötigt. Die Möglichkeiten aus den Bereichen Data Engineering und Machine Learning (ML) sind inzwischen so weit vorangeschritten, dass das Dark Genome mittels Artificial Intelligence (AI) immer weiter erschlossen werden kann. Wegweisende Modelle wie AlphaFold zur Strukturprädiktion von Proteinen und viele andere zeigen dies bereits deutlich. ProteinBERT, ESMFold oder Evo zeigen zudem, wie gegenwärtige Entwicklungen aus der natürlichen Sprachverarbeitung rund um den Ansatz des Self-Supervised Learnings und den Large Language Models (LLMs) ebenso als Treiber in der Genommedizin eingesetzt werden können.
Damit einhergehend ist eine der größten Herausforderungen der Genommedizin den enormen Datenmengen gerecht zu werden, die für die Genomanalyse und das Training von ML Modellen notwendig sind, denn ein einziges humanes Genom mit ca. 3,3 Milliarden Bausteinen benötigt ca. 3 Terabyte Speicher.
Um diesen Herausforderungen gerecht zu werden und neue Erkenntnisse in der Genommedizin zu schaffen haben wir in einem gemeinsamen Pilot-Projekt als Teil eines F&E Vorhabens mit dem Institut für Humangenetik und Genommedizin der Uniklinik RWTH Aachen (UKA), durch die Umsetzung einer cloudbasierten Software-as-a-Service (SaaS)-Plattform zur effizienten und umfänglicheren Genomauswertung, den Grundstein gelegt. Durch die Entwicklung und den Einsatz mehrerer ML-Services soll die Plattform zukünftig kontinuier
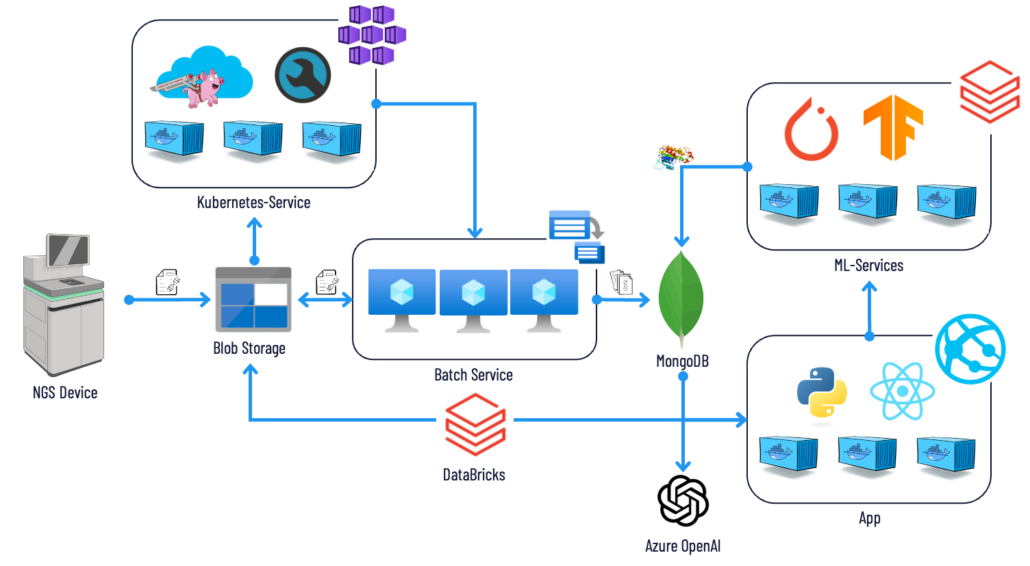
Einblicke in die Cloudarchitektur

Cromwell on Azure
Cromwell wurde vom Broad Institute entwickelt und ist ein Workflow-Management-System, das speziell für die Verarbeitung von Genomdaten entwickelt wurde. Die Software ermöglicht es, komplexe (Vorverarbeitungs-)Pipelines für die Analyse von genetischen Daten zu erstellen und auszuführen. Cromwell on Azure bietet die Möglichkeit zur flexiblen und skalierbaren Durchführung von Genom-Workflows, die eine hohe Rechenleistung erfordern.
Databricks
Mit seiner Lakehouse-Architektur bietet Databricks eine effektive Möglichkeit, große Datenmengen zu verwalten und zu analysieren. Die Plattform nutzt leistungsstarke Tools wie Apache Spark, um eine skalierbare und effiziente Umgebung für die Weiterverarbeitung der genetischen Daten bereitzustellen. Darüber hinaus dient Databricks der Erstellung und Orchestrierung von ETL-Pipelines. Gleichzeitig bietet die Plattform eine umfassende Umgebung für die Entwicklung, das Training und die Bereitstellung von ML-Modellen mit Tools wie TensorFlow und PyTorch.
Cosmos DB for MongoDB
Azure Cosmos DB ist eine NoSQL-Datenbank, die von Microsoft entwickelt wurde und verschiedene APIs unterstützt, einschließlich MongoDB. Die Verwendung von Cosmos DB als Datenbank bietet eine effektive Möglichkeit, Daten zu speichern und abzurufen und dabei die Skalierbarkeit und Flexibilität von Cosmos DB zu nutzen. Die Unterstützung der MongoDB-API erleichtert die Interaktion mit der Datenbank und ermöglicht Nutzern komplexe Abfragen und Analysen auf Basis der mittels Cromwell und Databricks verarbeiteten Daten.
Azure App Service
Die Verwendung von Azure App Service bietet eine umfassende Umgebung für die Entwicklung, Bereitstellung und Skalierung von Apps. Azure App Service wird verwendet, um eine App mit einer zielgruppenorientierten grafischen Benutzeroberfläche bereitzustellen, die als Schnittstelle zur zentralen Cosmos DB und zur Nutzung der ML-Services dient. Dies ermöglicht es Benutzern, Varianten in den bisher wenig beachteten Non-Coding-Regionen (Dark Genome) zu identifizieren und neue Erkenntnisse zu gewinnen.
Fazit
Wir sind davon überzeugt, dass unsere interdisziplinäre Zusammenarbeit aus Bereichen Data Engineering, Data Science sowie der Humangenetik und Genommedizin dazu beitragen wird, das Verständnis für seltene Erkrankungen und die Lebensqualität von Menschen mit diesen Erkrankungen zu verbessern. Wir freuen uns weiterhin auf diese tolle Zusammenarbeit und sind gespannt auf die neuen Erkenntnisse, die wir, getragen durch die Entwicklung von neuen und der Anwendung von bestehenden Machine Learning Modellen, gewinnen werden.
Autor
Martin Danner, Data Scientist bei scieneers GmbH
martin.danner@scieneers.de






 dpunkt.verlag GmbH
dpunkt.verlag GmbH